IVF_PQ Explained
사내에서 사용하는 유사 이미지 검색 서버를 개선하는 작업을 진행하던 중, IVF_PQ 에 대해 정확히 이해하고 사용하고 싶어 여러 문서들을 찾아보았으나 100% 이해가 안되어서 논문을 읽고 여러 문서들을 참고하여 정리하였습니다. (편의상 아래 내용부터는 경어를 사용하지 않습니다.)
기존에 FAISS 를 사용하고 있는데, 샤딩을 라이브러리 단에서 지원하지 않아 분산 처리를 하기 어려운 문제가 있다. 특히, 분산 시스템이 늘상 그렇듯이… Insert 가 빈번하게 일어나는 경우 여러 고려가 필요한데 때마침 Milvus 라는 Distributed Vector Similarity Search 오픈소스 프로젝트가 있어서, 이를 검토하고 있음.
다만 Milvus는 분산 환경을 서포팅하기 위해 인덱스를 개별 파일 단위로 쪼개서 여러개를 생성함. 반면 Faiss는 싱글 인덱스이다. 기존 시스템을 Milvus로 옮기려면 이를 위한 Indexing 설계 및 후처리가 필요한데, 인덱싱이 어떻게 이뤄지는지 모르고는 당연히 잘 처리할 수 없다. (애초에 제대로 모르면 그게 필요하다는 사실조차 모를 확률이 높다…)
NN? ANN?
IVF_PQ 에 대해 말하기 전 잠시 ANN 에 대해 짚고 넘어가자.
ANN 은 Approximate Neareast Neighborhood Search (not artifitial neural network ㅋㅋ) 의 약자로 검색 퀄리티랑 [속도(==연산량), 메모리] 사이의 trade-off 를 하는 검색 기법이다.
NN의 예시
NN 의 예시는 (not Approximate!) 다음과 같다.
- Brute-force: 말 그대로 모든 벡터와 다 비교해서 찾는 것. 가장 정확하지만 연산량이 많음
- 검색 연산량: O(N)
- 메모리: O(1)
- 하나의 file system 에 벡터를 저장하고 순차적으로 읽는다 가정
- K-means: 인덱싱 시에 전체 벡터 공간을
nlist개의 그룹으로 나눠서 해당 그룹에서만 찾는 전략- 먼저 학습 단계가 필요함. 각 그룹의 중심 벡터, 즉
centroid를 학습 단계에서 구해서 저장 (그룹 내 모든 벡터와 distance 차의 합이 최소) - 검색 시에는 쿼리 벡터와 거리가 개별 그룹의
centroid사이의 거리를 계산 후 가장 가까운 그룹을 찾아 그룹 내 벡터와 연산하여 top-k 개의 벡터를 뽑음 - 검색 연산량:
O(d * (nlist + n/nlist))- 전체 데이터 개수가 n, 각 벡터의 차원이 D
- 검색 시간이 n 에 비례함!!! (nlist 는 상수로 고정)
- 메모리:
O(nlist * D + n * D)centroid를 메모리에 저장, 모든 벡터를 메모리에 저장
- 어떻게 되는지 설명하는 짤
- 먼저 학습 단계가 필요함. 각 그룹의 중심 벡터, 즉
위에서 언급한 K-means은 가장 naive 한 방식이고, 실제로는 학습 단계에서 tree 를 구성하는 등의 방법이 있음. 자세히 살펴보진 않았지만 tree 라면 Insert 에서 불리하고, leaf 이외의 개별 노드를 다 메모리에 올리거나 하는 등의 과정이 필요할텐데… 현실적으로 사용이 어려울 것 같음 (실제로 안찾아봐서 정확하진 않으나 생각해보면…)
차원의 저주
위의 과정은 거의 O(N) 이다. (nlist 와 같은 hyperparam 은 상수라 무시)
Million 스케일에서도 힘든데, Billion 은 당연히 어렵다. 위와 같은 이유로 대용량 처리 시에는 연산량이나 메모리 문제로 ANN 이 사실상 필수적
ANN
차원의 저주를 피하기 위해 적절한 trade-off 를 통해 검색 시 연산량을 O(n) 에서 O(n^x) (x < 1) 로 줄임 (또는 log…)
- Tree 기반: HNSW, … ← 여기서는 설명하지 않음
- IVF 기반: IVF_PQ, … (여기서 사용할 것들)
IVF_PQ
- IVF
- Inverted File
- 위의 K-means 에서, 모든 벡터를 메모리에 올리지 않기 위해 최적화할 수 있음. 각
centroid에 file-pointer 를 mapping 해서 (inverted) 해당 위치에 centroid 가 속하는 그룹의 모든 벡터를 append. - 이 경우 검색 시에 어느 그룹인지를 알면 해당 그룹의 파일만 읽어서 처리하는 것이 가능해져서 메모리 사용량을 줄일 수 있음
- 그러나 여전히
centroid는 메모리에 저장해야하므로, Milvus 와 같은 분산 인덱스에서 개수가 많아지면 큰 오버헤드로 작용- ex) D=1536, nlist=1024 의 경우 필요한 메모리는
1k * 1536 * 4B = 6MB인데 분산 인덱스 개수가 10240개라면 centroid 를 저장하는데만 60GB 를 소요
- ex) D=1536, nlist=1024 의 경우 필요한 메모리는
- PQ
- Product Quantization
- IVF 등으로 검색 개수를 줄이더라도 검색시 연산량이 O(n)이고, 해당 그룹 내 벡터를 메모리에 올려야함!
- 이를 최적화하기 위해 아래와 같은 작업을 수행
- 모든 벡터를 m 개의 subvector 로 쪼개고, 각 sub-vector 들에 대해 K-means 을 수행
- sub-vector 들이 속한 group 에 id 를 부여하는데, d bits 로 표현한다면 sub-vector 마다 2^d 개의 id 가 생김
- sub-vector 의 Cartesian 곱으로 각 벡터가 2^d^m 개의 group 으로 분리됨
- ex) [0.0, 1.0, … , 1535.0] ⇒ [5, 243, 15, … , 47] 과 같은 sub-vector id 들의 튜플로 표현 가능
- e.g.) 앞의 벡터는 1536차원, 뒤의 튜플은 48차원
- ex) [0.0, 1.0, … , 1535.0] ⇒ [5, 243, 15, … , 47] 과 같은 sub-vector id 들의 튜플로 표현 가능
- 그러면서도 메모리 사용량은 굉장히 적음
D' ← D/m이라고 하면 m 개의 sub-vector 들에 대해 2^d 개의 centroid 만 저장하면 되므로, 전체 centroid 를 저장하는데는m * D' * 2^d * 4Bytes ⇒ D * 2^d * 4Bytes.- 각 벡터를 표현하는데는 sub-vector 의 id 만 저장하면 되므로
m * d bits- m=32, d=8 인 경우 한 벡터당 겨우 32Bytes !
- e.g.) D=1536, m=32, d=8 인 경우 표현 가능한 group 은 2^40 인데 사용하는 메모리 량은
1536 * 2^8 * 4Bytes ⇒ 1.5MB로 가능- K-means 이었다면
1536 * 2^40 * 4Bytes => 6TB
- K-means 이었다면
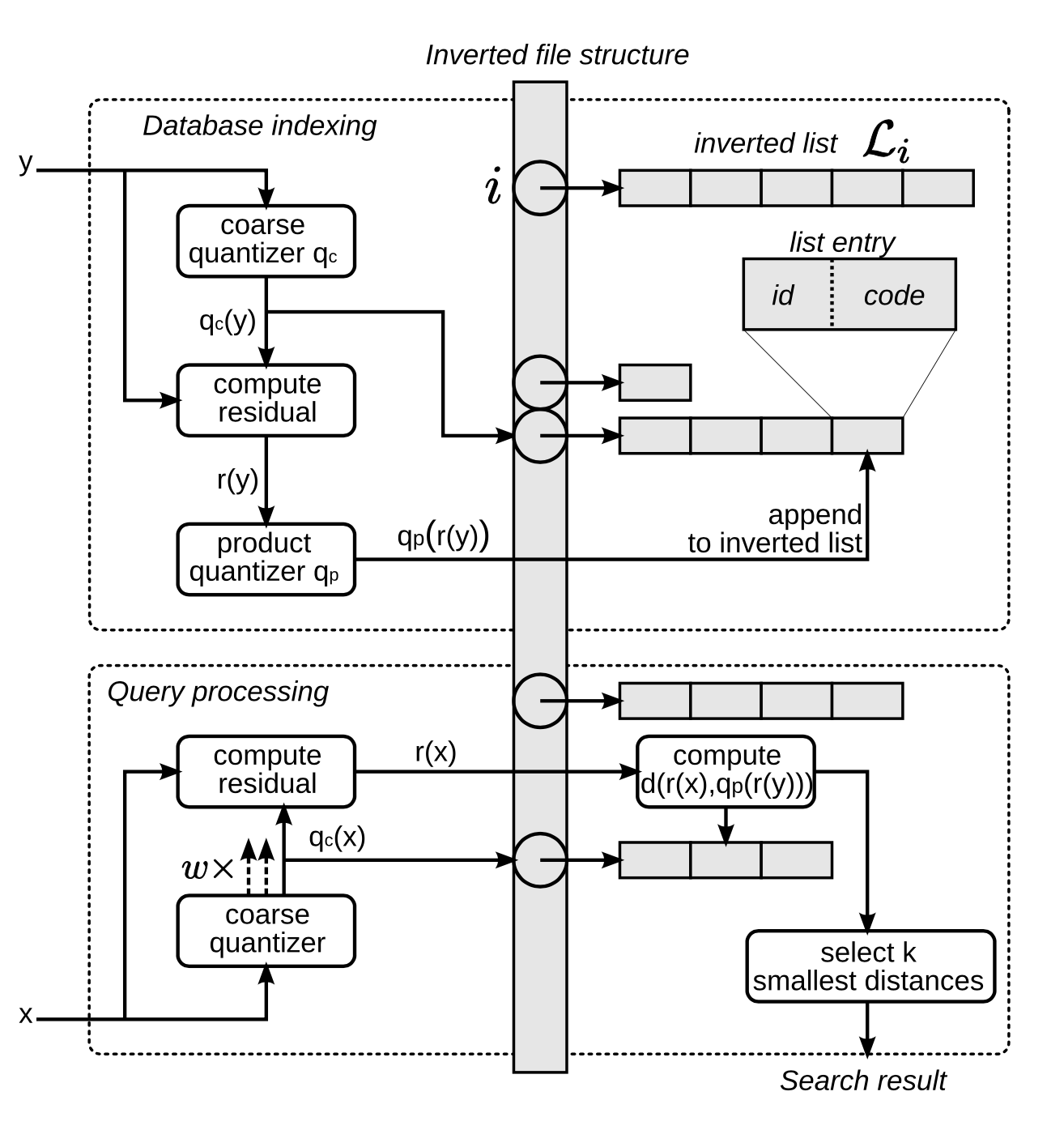
- IVF_PQ
- 위의 두개를 합한 형태인데 아래 그림으로 표현 가능
- coarse quantizer 로 먼저 K-means 을 수행하는데, 원래 차원을 그대로 유지하면서 nlist 개 만큼의 그룹으로 나눔
- 아래 그림과 같이 r(y) 에서 PQ 를 계산해서, 각 sub-vector 의 centroid id list (== code) 를 ivf 에 추가
- 검색 시에는 다음과 같이 처리 (여기서는 논문 상의 ADC, 비대칭 계산이에요.)
- 우선 쿼리 벡터와 ivf 에서 사용된 centroid 들과 거리 계산 후 정렬
top-nprobe개의 그룹에 대해 (여기서는 w) r(x) 를 구하고, 해당 r(x) 와 모든 sub-vector centroid 와의 거리를 계산한 후, distance table 을 생성- sub-vector centroid 는 2^d * m 개이고, r(x) 를 m 개로 나눈 뒤 각각에 대해 2^d 개와 거리 계산
- D=1536, d=8, m=32 라고 하면 실제 계산은
32 * (1536/32 * 256)⇒ 금방 처리 가능
- D=1536, d=8, m=32 라고 하면 실제 계산은
- sub-vector centroid 는 2^d * m 개이고, r(x) 를 m 개로 나눈 뒤 각각에 대해 2^d 개와 거리 계산
- ivf 내의 각 벡터들의 sub-vector id 튜플 들에 대해, 위에서 만든 table 을 룩업해서 더하여 거리를 계산
- 이후 top-k 개 만큼 추출
- 위의 두개를 합한 형태인데 아래 그림으로 표현 가능
Faiss vs Milvus
Faiss
모든 벡터를 하나의 인덱스로 만듦
- IVF 를 전체에 대해서 딱 한번만 만들어서 오버헤드가 작음
- 하나의 인덱스를 쪼개기 어려운 구조라 분산 처리가 어려움
Milvus
초기에 지정한 파일 사이즈를 넘기면 강제로 분리해서 인덱스를 새로 만듦
- 개별 인덱스에 대해 IVF (coarse quantizer) 를 만드므로 메모리 소모가 아주 큼
- 분산처리가 쉬움
- 같은 환경이라면 Faiss 보다 느림. 이유는 분산된 인덱스에 대해 각각 top-k 개 만큼 다 뽑은 다음에 합쳐야함
- 단, Faiss 는 감당 가능한 수치를 넘었을 때는 샤딩이 네이티브로 지원되지 않으므로 직접 구성해야함
- 스케일 아웃으로 Milvus 를 구성 시 Faiss 보다 훨씬 대용량을 안정적으로 서빙 가능
Milvus의 경우 필요한 메모리를 계산해주는 툴이 있어서 sizing-tool을 참고해서 메모리 사용량을 계산할 수 있음.
Seg file size 를 줄이면 파일이 쪼개져서 인덱스가 여러개 만들어지는데, 오버헤드가 얼마나 큰지 확인해볼 수 있음.
다음과 같은 (#Vectors, Dimesion, nlist, m, Segment file size) 순서로 Param 을 표현할 때, 대략 다음과 같음.
(100M, 1536, 1264, 32, 1024)=> 31GB(100M, 1536, 1789, 32, 1024)=> 22GB(100M, 1536, 2529, 32, 1024)=> 17GB
nlist 의 경우 4 * sqrt(#vectors in segment file) 이 권장사항이므로 이를 통해 계산. 1GB에서 대략 10만개의 Vector 가 담김
유사 이미지 검색 in Milvus
Faiss 의 경우 싱글 인덱스이므로 위의 IVF_PQ 방식을 통해 top-k 를 구하면 global optima 에 근사함
Milvus 의 경우 멀티 인덱스이므로 가장 유사한 top-k 개의 이미지를 naive 하게 구하면 안되는데, Milvus 처럼 쪼개진 인덱스의 경우 각 인덱스에 대해 PQ 가 다르게 적용되어 PQ centroid 가 조금씩 달라서 오차가 누적되기 때문.
일반적으로 싱글 인덱스에서는 거의 비슷한 벡터의 경우 sub-vector 중에 일부분의 id 만 달라서 top-1 과 id 가 아주 조금 다르겠지만, 다른 인덱스의 경우 개별 sub-vector 의 centroid 값이 서로 달라서 오차가 누적됨
- Faiss 의 경우
- 거의 유사한 이미지 a, b가 있다고 가정하면 a,b 가 각각 PQ 된 id list 는 다음과 같은 식
- a: [0,1,2,3,…,47], b: [0,1,4,3,…,47]
- 쿼리 벡터 q 에 대해 d(q,a), d(q,b) 는 저 빨간색 centroid 차이 만큼만 나므로 두 거리의 차가 작게 나옴
- 거의 유사한 이미지 a, b가 있다고 가정하면 a,b 가 각각 PQ 된 id list 는 다음과 같은 식
- Milvus 의 경우
- 거의 유사한 이미지 a, b가 있는데, 각각 다른 인덱스에 있다고 가정하면 a,b 가 각각 PQ 된 id list 는 다음과 같은 식
- a: [0,1,2,3,…,47], b: [10,11,14,13,…,147]
- 또한, 각각의 id 에 해당하는 centroid 의 경우도 인덱스마다 다르게 계산되므로, 아주 가깝게 나오겠지만 완전히 일치하지는 않음
- 쿼리 벡터 q 에 대해 d(q,a), d(q,b) 는 모든 centroid 의 차이만큼 누적되므로 거리가 크게 나옴 ⇒ 실험 결과 거의 유사한 이미지더라도 거리 차이가 꽤 큼
- 거리 차가 0.1, 0.01 이런식으로 나오는게 아니라 거리 차가 5, 10 이런식
- 다만, 적절한 파라미터를 줬다는 가정하에 랭킹은 유지될 확률이 큼
- 거의 유사한 이미지 a, b가 있는데, 각각 다른 인덱스에 있다고 가정하면 a,b 가 각각 PQ 된 id list 는 다음과 같은 식
- 가장 가까운 image_id 리스트를 받고, 각 image_id 를 통해 원본 벡터를 읽어서 쿼리 벡터와 다시 거리를 구하는 Post-processing 를 해야함
Future Works
현재 Milvus를 실험 중인데 공식 문서들과 medium 내용들을 보며 많은 부분을 정리하고, 노하우를 쌓아가고 있음. Future Works 로 아래의 글들을 추가해서 쓸 예정.
혼자 공부하면서 정리해나간 글인데, 혹여 틀린 부분이나 글을 잘 못써서 이해가 잘 안가는 부분이 있을 수 있어서 언제든지 같이 얘기해보는 것 환영입니다.
